Библиометрические инструменты в помощь исследователю
ПОИСК
Итак, вы заинтересовались новой научной темой и открыли поисковую систему Scopus. Что определяет ее работу? Можно назвать два важных компонента – механизм поиска (правила обращения к определенным полям базы данных) и качество контента (однородность наполнения и частота ошибок).
Возможности поисковой системы Scopus опубликованы на сайте Elsevier в виде описания правил обращения к базе (команды, операторы, поля), поэтому мы не будем их обсуждать. Вместо этого мы поговорим о том, как влияют особенности описания документов в Scopus на качество поиска.
Поисковая машина обращается к описаниям документов, которые могут ощутимо различаться по наполнению отдельных полей.Так, если журнал индексируется не только в Scopus, но и в базах данных Reaxys, Embase или Compendex, также принадлежащих Elsevier, то из последних в Scopus попадают словарные термины (index terms) и идентификаторы химических соединений (chemical names). На рисунке представлен фрагмент описания публикации из журнала Angewandte Chemie - International Edition (10.1002/anie.201305489), в котором помимо названия статьи, реферата и авторских ключевых слов присутствуют термины из нескольких «словарей» (EMTREE, MeSH и др.) и CAS номера химических соединений.
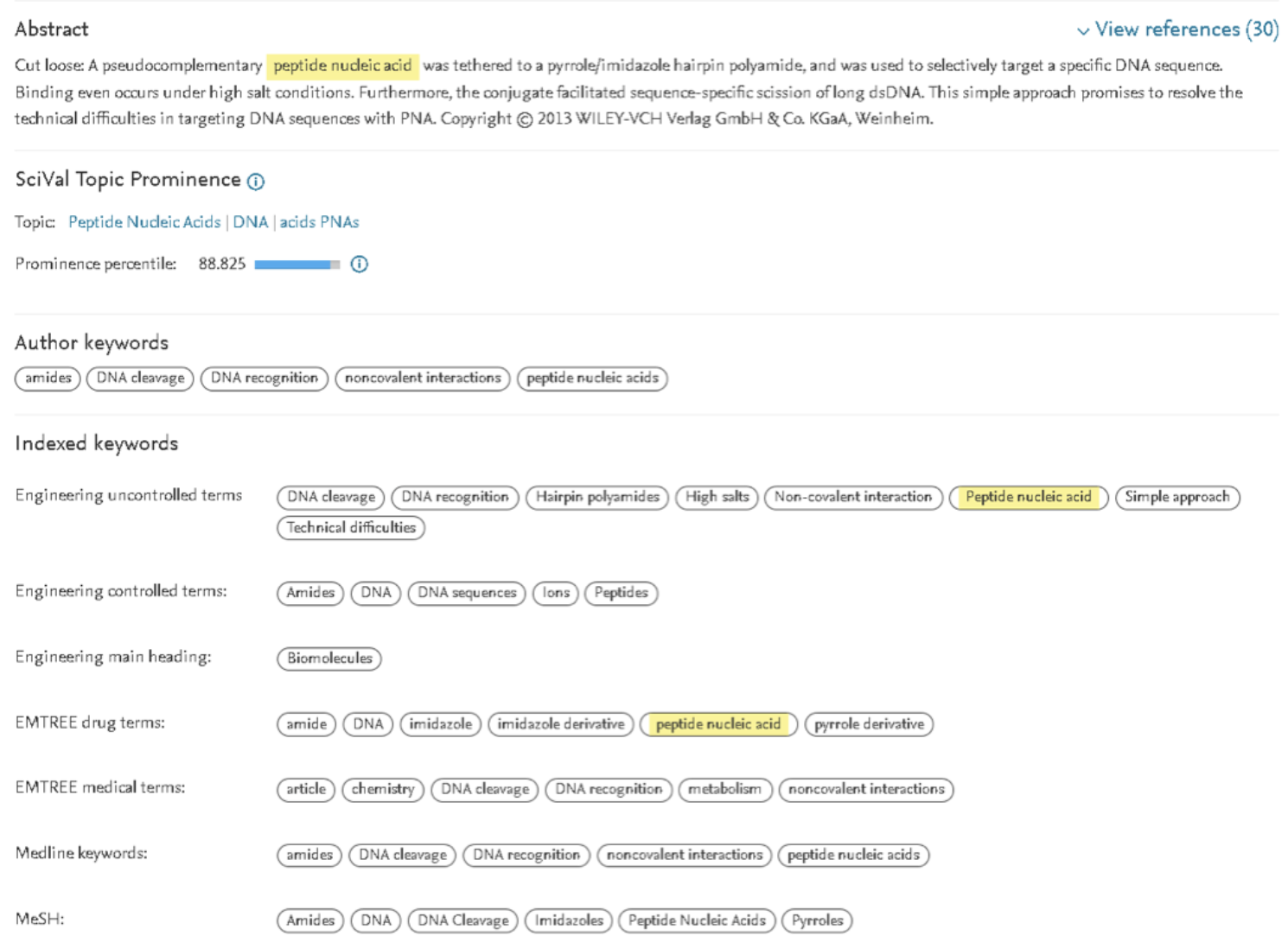
Добавление этих унифицированных терминов, с одной стороны, облегчает поисковую работу исследователя, поскольку вместо угадывания авторских ключевых слов (синонимов, вариаций написаний, откровенных ошибок), достаточно использовать единственный «правильный» термин или название соединения. Но вместе с тем, появление в описании статей новых терминов повышает вероятность нахождения статьи по другим, менее релевантным поисковым запросам. Так, вышеупомянутая публикация из-за добавления более общих терминов будет обнаруживаться в результатах поиска metabolism AND peptides.
Таким образом, добавление словарных терминов, с одной стороны, значительно повышает чувствительность поиска, с другой – снижает его специфичность.
Необходимо также учитывать, что Scopus охватывает больше изданий, чем перечисленные базы данных, поэтому некоторые статьи там не содержат в своем описании ни index terms, ни CAS идентификаторов.
И, наконец, в Scopus (как в любой реферативной базе данных) можно встретить документы, в описании которых отсутствует реферат и/или авторские ключевые слова, таким образом, поиск будет осуществляться только по названию документа.
Резюмируем. Выбор поисковой стратегии – это всегда компромисс между чувствительностью и специфичностью, выбор между усилиями, необходимыми для обнаружения документов с недостаточным описанием, и теми, которые потребуются для отбора малорелевантных статей из результатов поиска.
ЧАСТЬ 2. КОНТЕНТ
В предыдущей части мы говорили том, как влияет качество контента на работу поисковой системы и что следует учитывать исследователю. В этот раз речь пойдет об особенностях анализа ключевых слова в интерфейсе поискового сервиса. Представим задачу: молодому исследователю предстоит сделать обзор литературы по пептид-нуклеиновым кислотам, о которых он знает лишь самые общие сведения.
Поскольку тема сформулирована через название объекта исследования (название химического соединения), то стратегия поиска сводится к перебору вариантов названия: peptide-nuclic acids ИЛИ peptide nucleic acids ИЛИ PNAs. Принимая во внимание, что Scopus не обращает внимания на дефисы, а аббревиатура слишком короткая, поисковой запрос TITLE-ABS-KEY ("peptide-nucleic acid") будем считать достаточным.

После просмотра первых страниц с результатами поиска исследователь скорее всего заметит, что PNA не только сами по себе являются объектом исследования, но также имеют несколько вариантов прикладных приложений. И возникает естественный вопрос: можно ли выделить отдельные исследовательские направления?
Что предлагает нам Scopus?
Вариант #1. Scopus позволяет просматривать в панели фильтров список ключевых слов и выгружать их через функцию Export Refine.

Но можно назвать несколько причин не следовать по этому пути:
а) просматривать и выгружать таким образом можно не все, а только первые 100 - 200 терминов. Поскольку словарных терминов (index terms) больше, чем авторских слов, в видимой части списка присутствует много общих терминов – Human, Chemistry, Nucleic Acids и т.п.
б) применение фильтра для нового ключевого слова равносильно поиску его по полям Author Keywords и Index Terms, но ключевое слово может встречаться не только в этих полях, но также в названии статей или рефератах.
Проиллюстрируем сказанное, выбрав дополнительный термин "Epidermal Growth Factor Receptor" и нажав Limit to:
Поисковой запрос TITLE-ABS-KEY ("peptide-nucleic acid") AND (LIMIT-TO(EXACTKEYWORD, "Epidermal Growth Factor Receptor" )) возвращает 115 результатов.
Если включим этот термин в поисковой запрос по стандартным полям, то Scopus TITLE-ABS-KEY ("peptide-nucleic acid" ) AND TITLE-ABS-KEY ( "Epidermal Growth Factor Receptor"), вернет 166 документов, т.е. в полтора раза больше результатов.
в) применение фильтра пропустит статьи, в которых используются сокращенные формы термина – EGFR или EGF receptor.
Таким образом, исследователь стоит перед задачей выбора релевантных терминов из длинного списка, который он не в состоянии просмотреть. С каждым новым поиском (добавлением ключевого слова) объем работы исследователя увеличивается, поскольку вынуждает переходить в интерфейс поиска документов, сохранять результаты в списки и т.д.
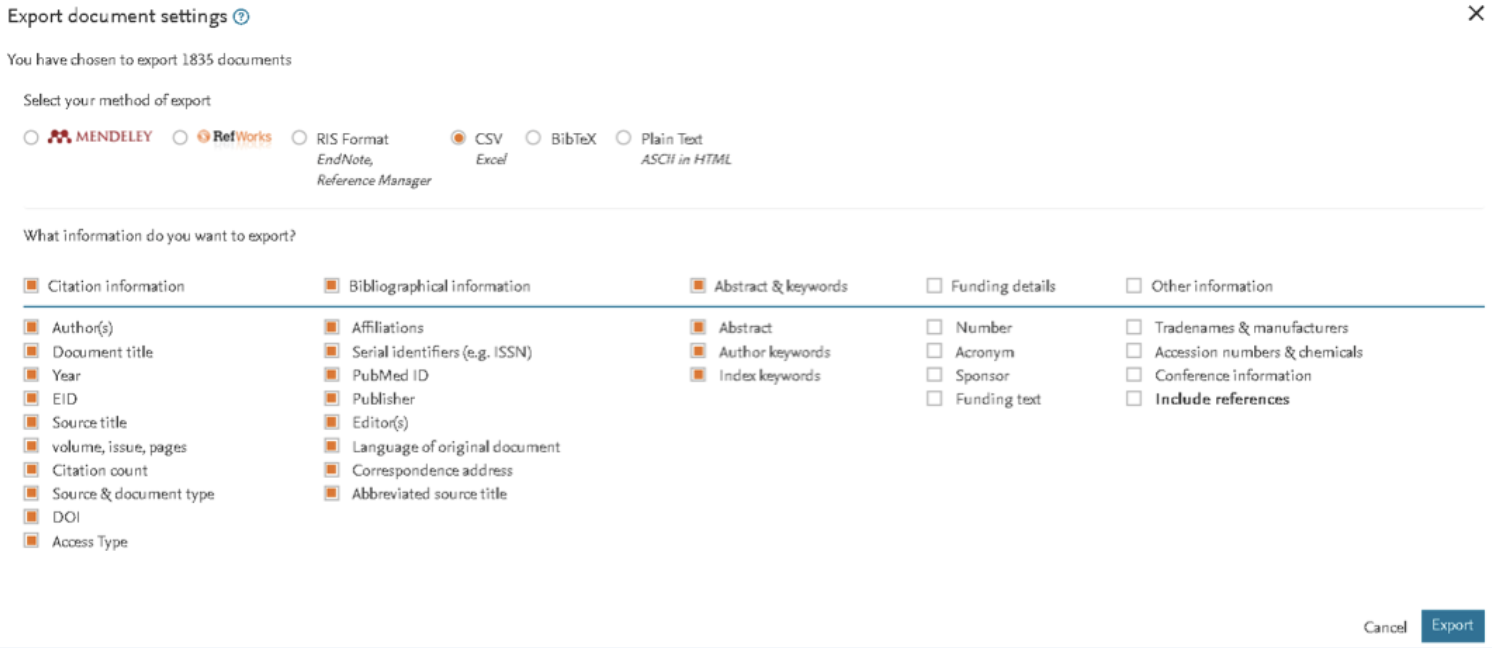
Вариант #2– Scopus позволяет выгрузить все найденные результаты в формате CSV, в том числе и ключевые слова, что позволит исследователю анализировать контекст публикаций оффлайн.
Выгрузим публикации о peptide nucleic acids, опубликованные с 2008 года по настоящее время в виде публикаций типа Article (или Article in Press).
TITLE-ABS-KEY("peptide-nucleic acid" ) AND PUBYEAR > 2007 AND (LIMIT-TO(DOCTYPE ,"ar" ) OR LIMIT-TO(DOCTYPE ,"ip" )) вернул 1835 документов, описание которых можно выгрузить из Scopus за один раз (лимит - 2000 документов, при необходимости выгрузки для большего числа результатов, их следует разбивать на группы, например, фильтрацией по годам). Сохраняем файл в формате csv.
Часть 3.
В предыдущих постах мы начали анализ научных тем, связанных с пептид-нуклеиновыми кислотами, и пришли к выводу, что анализ ключевых слов в интерфейсе Scopus для этой задачи не вполне удобен. Эту задачу мы будем решать с помощью программы VOSviewer.
Здесь сделаем небольшую, но значимую ремарку – мы не ставили перед собой задачу написать руководство по работе с программой, поэтому не каждый шаг будет сопровождаться соответствующим скриншотом или разъяснением. По этой же причине мы будем обсуждать не все возможности программы, а лишь те, которые нам могут помочь для решения конкретной задачи.
VOSviewer разработан сотрудниками Центра исследований науки и технологий (CWTS) Лейденского университета (https://www.cwts.nl). Программа позволяет работать с данными из разных источников (WoS, Scopus, Dimensions,CrossRef, Medline), регулярно обновляется, распространяется бесплатно и не требует инсталляции.
Скачиваем с сайта http://www.vosviewer.com/ подходящую версию, распаковываем архив и, собственно, всё.

Возможность выбора между авторскими ключевыми словами и словарными терминами, как мы увидим позднее, является несомненным достоинством программы.
Шаг 1. запускаем файл VOSviewer.exe
Шаг 2. нажимаем Create
Шаг3. выбираемCreate a map based on bibliographic data
Шаг 4. на вкладке Scopus указываем расположение файла CSV(см. предыдущий пост)
Шаг5. выбираем в соответствующих разделах Co-occurrence, All keywords, Full counting.
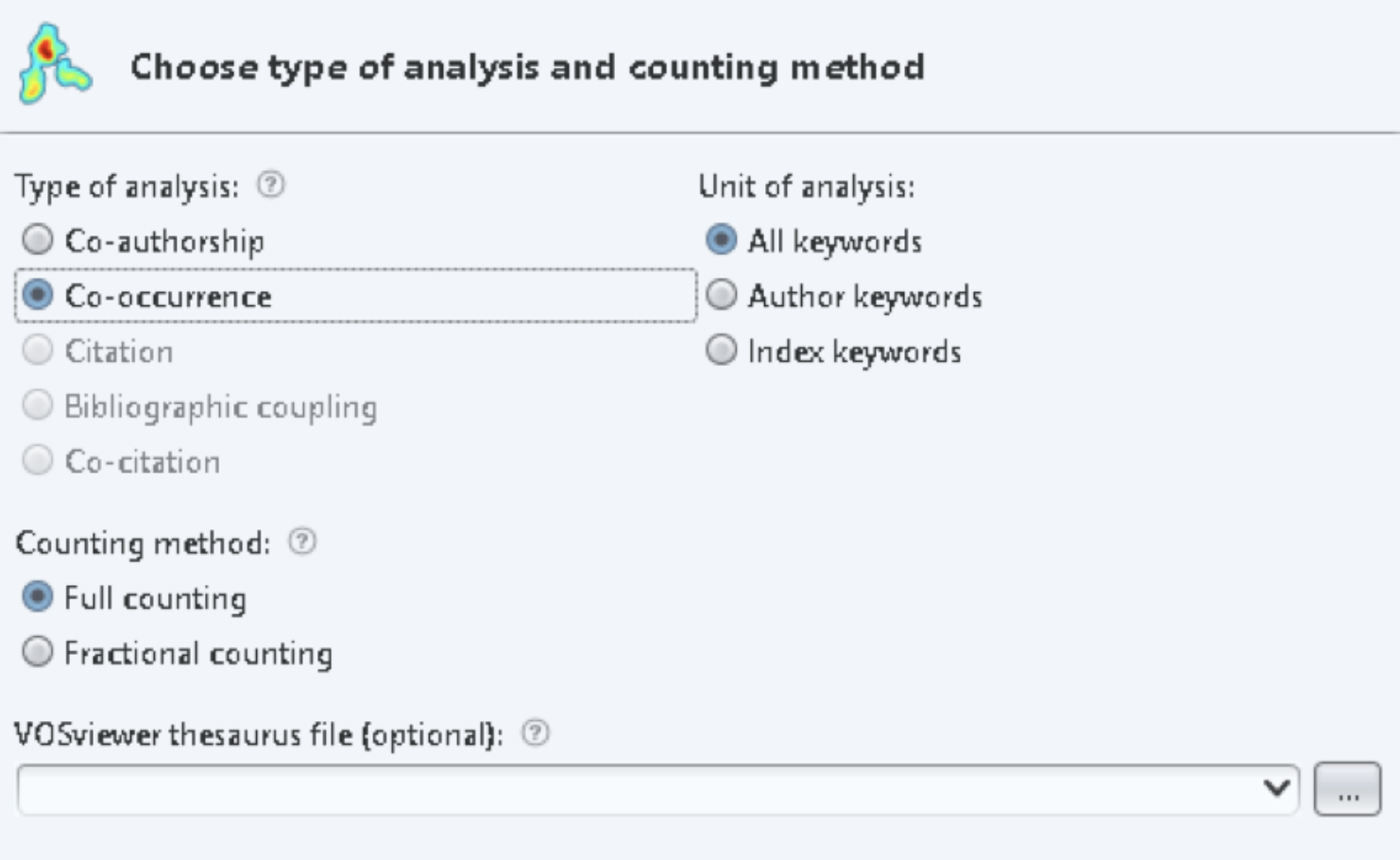
Шаг 6. Программа анализирует файл и предлагает ввести порог встречаемости ключевых слов для удаления из анализа совсем редких терминов.
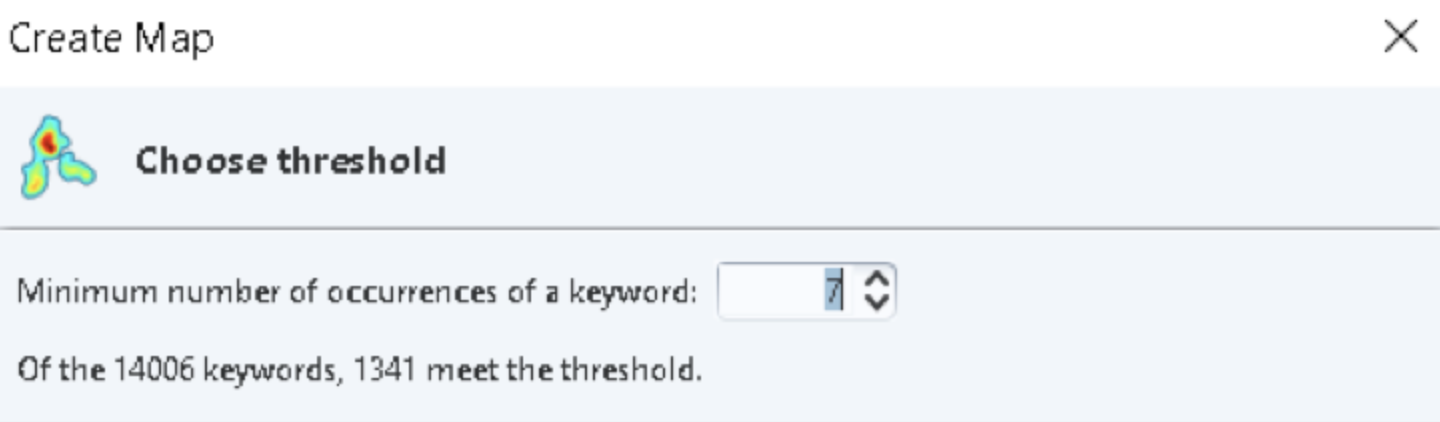
В нашем случае найдено 14006 ключевых слов – такое количество точек на экране сделает визуальную оценку невозможной. Попробуем ограничиться анализом только тех ключевых слов, которые встречаются минимум 7 раз, что сократит количество точек до 1341. Удаляя редкие термины, мы «снижаем уникальность» описания каждой публикации, или выражаясь фигурально, выбираем для микроскопа более слабые увеличительные стекла.
Шаг 7. Программа предложит сократить количество терминов, для которых будет проводиться расчет, до одной тысячи. Откажемся от этой автоматической настройки и выберем полное количество (1341).
Шаг 8. На последнем этапе VOSviewer предлагает в последний раз взглянуть на список терминов с возможностью отредактировать его вручную. Для нас это важный этап, поскольку он дает возможность удалить слишком общие термины – такие как human, humans, priority journal, article, male, femaleи т.д.. Удаляя малоинформативные термины, мы повышаем специфичность связей между публикациями, что должно оказывать положительный эффект на результат кластеризации.
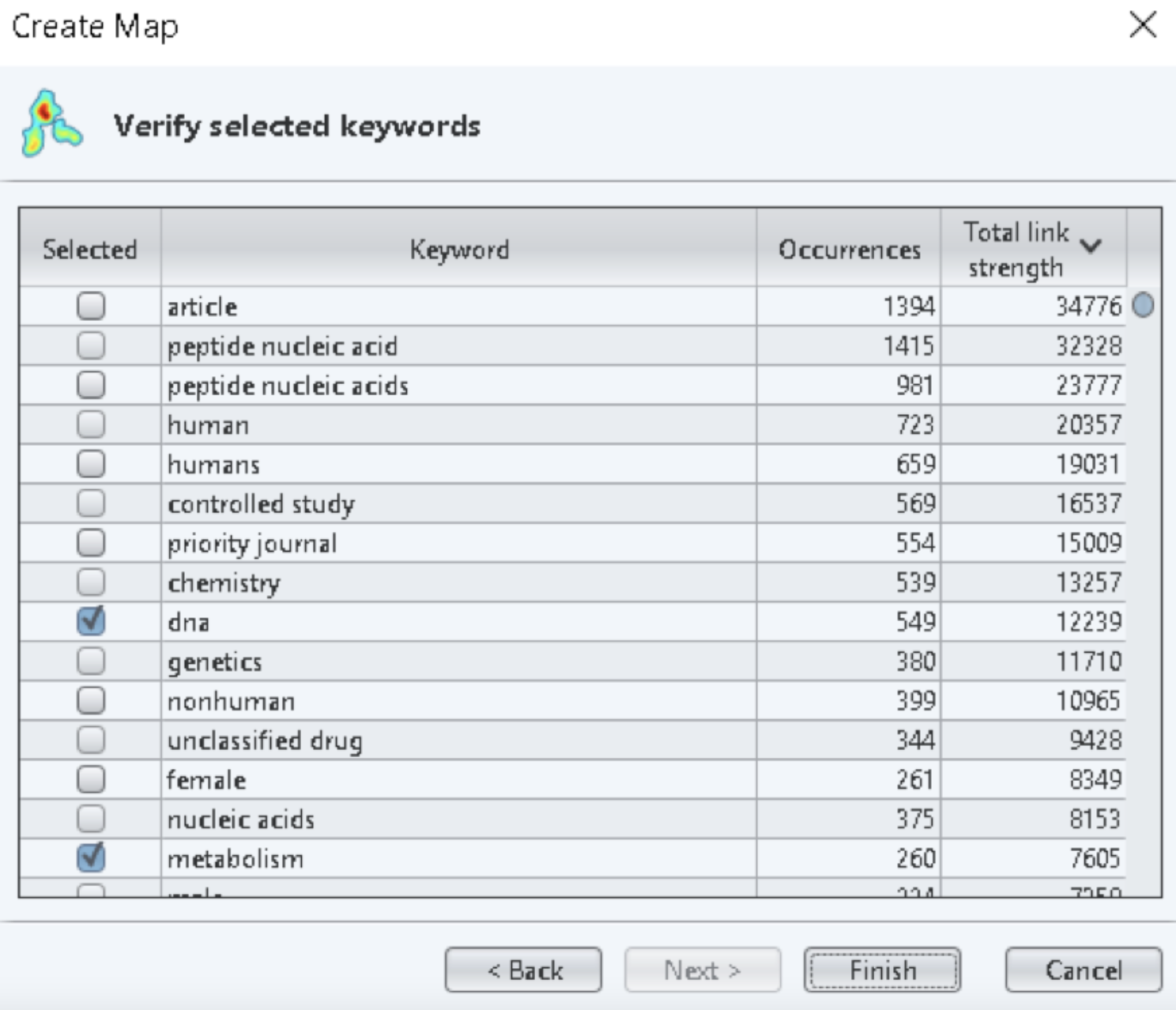
Просматривая ключевые слова, мы можем заметить различные варианты написания одного и того же термина, например, human и humans, preschoolchild и child, preschool, retrospective study и retrospective studies. К сожалению, программа воспринимает их, как разные термины, что ухудшает результаты кластеризации. Именно для этой цели в VOSviewer предусмотрена возможность использования словаря (см. далее).
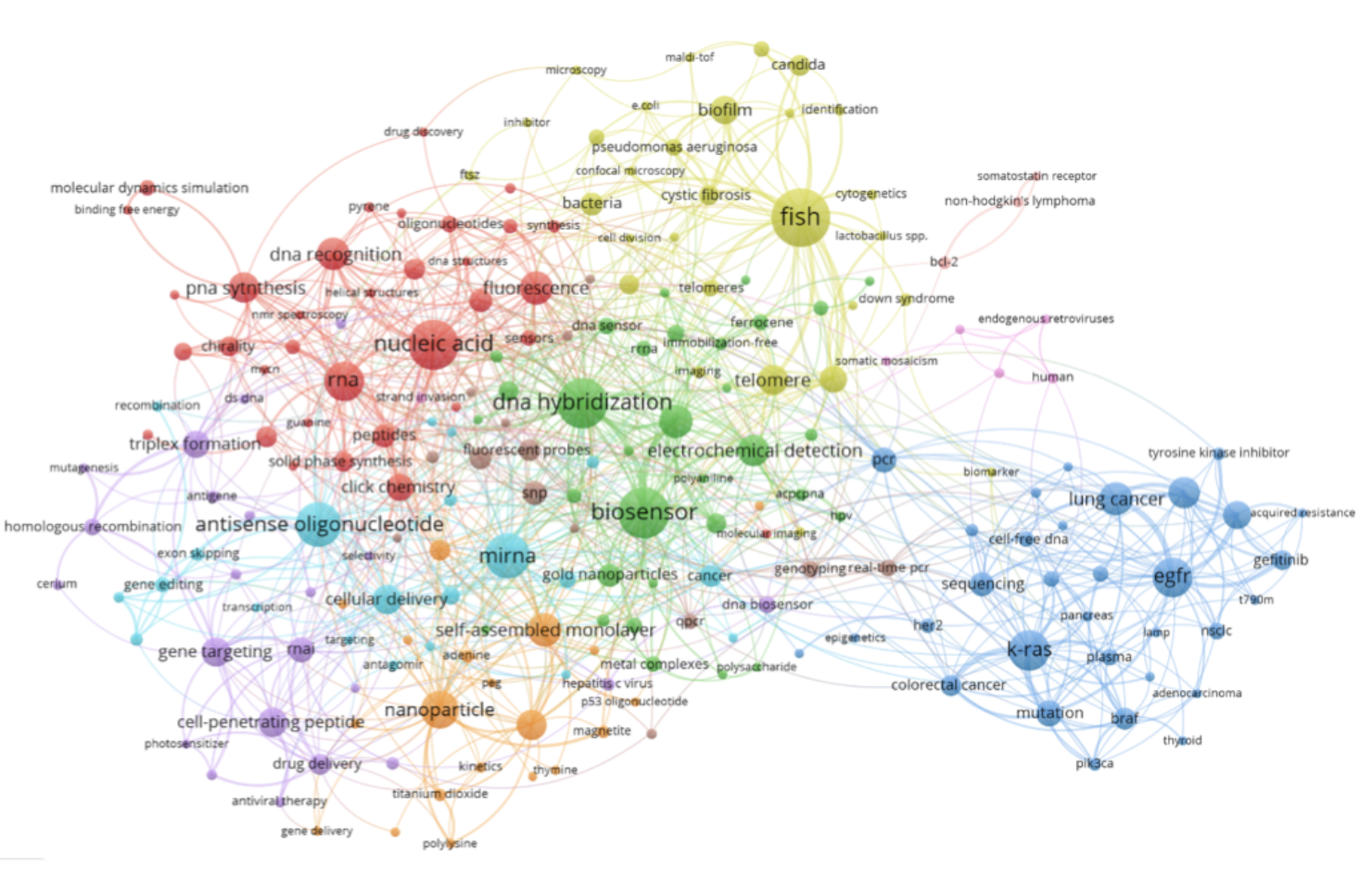
Шаг 9. Нажимаем Finish, ждем результат и любуемся получившейся картиной.
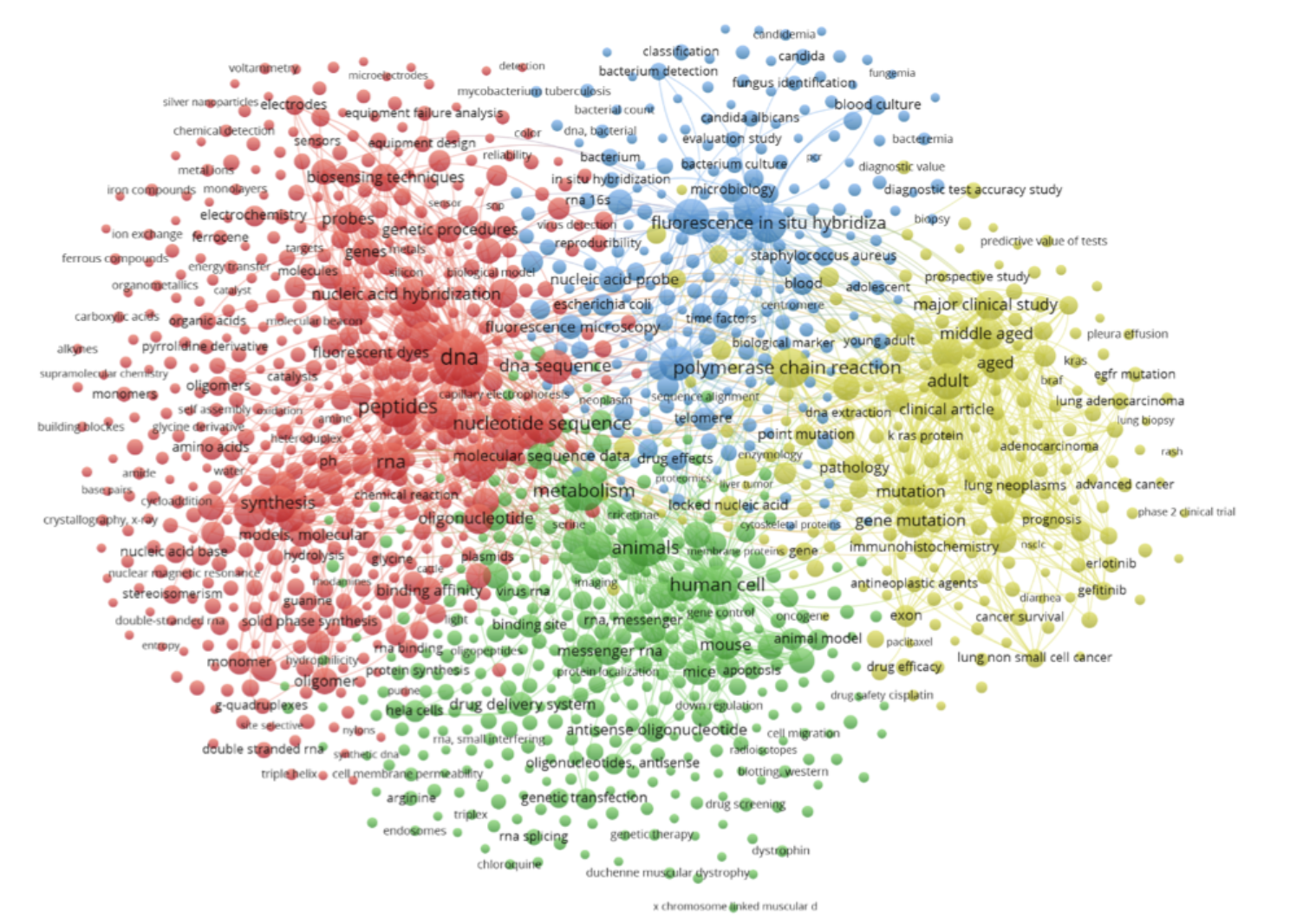
Программа по умолчанию выделила несколько крупных зон, в которых угадываются действительно разные направления исследований (красная и синяя зоны – диагностика и чипы, зеленая – регуляция экспрессии, желтая – клинические испытания).
Но вместе с этим, на картине по-прежнему видны общие и малоинформативные термины, которые вряд ли помогают разделению тем – например, термины adult, middleage, agedможно удалить, как и human, male, female(конечно, если мы уверены в том, что PNA не имеет различного использования в разных возрастно-половых группах).
В эту же группу кандидатов на удаление можно отнести словарные термины –gene, mutation, peptides, aminoacids, oligomer, pathologyи т.п., они описывают объект, а не способ его исследования или применения, тогда как нас интересует последнее.
Одним из неудобств системы VosViewerявляется невозможность редактирования списка узлов графа (в нашем случае это термины) on the fly, то есть прямо на экране, без повторной загрузки. Если мы решили удалить или объединить несколько узлов (например, синонимов), нам потребуется пройти все этапы загрузки файла с самого начала. Однако, это неудобство не следует воспринимать как недостаток программы, потому что ручная коррекция узлов графа – это верный путь к получению результатов, которые потом невозможно воспроизвести. Мы вернемся к вопросу, как следует вносить изменения, когда будем рассматривать работу с тезаурусом.
Попробуем теперь взглянуть на «лексическое пространство» исследований PNA, взяв только авторские ключевые слова (Author Keywords) и установив порог в минимум 3 упоминания (328 слов). Поскольку издатели обычно ограничивают количество ключевых слов, которые автор может указывать при подаче статьи, последние стремятся использовать действительно специфические термины.
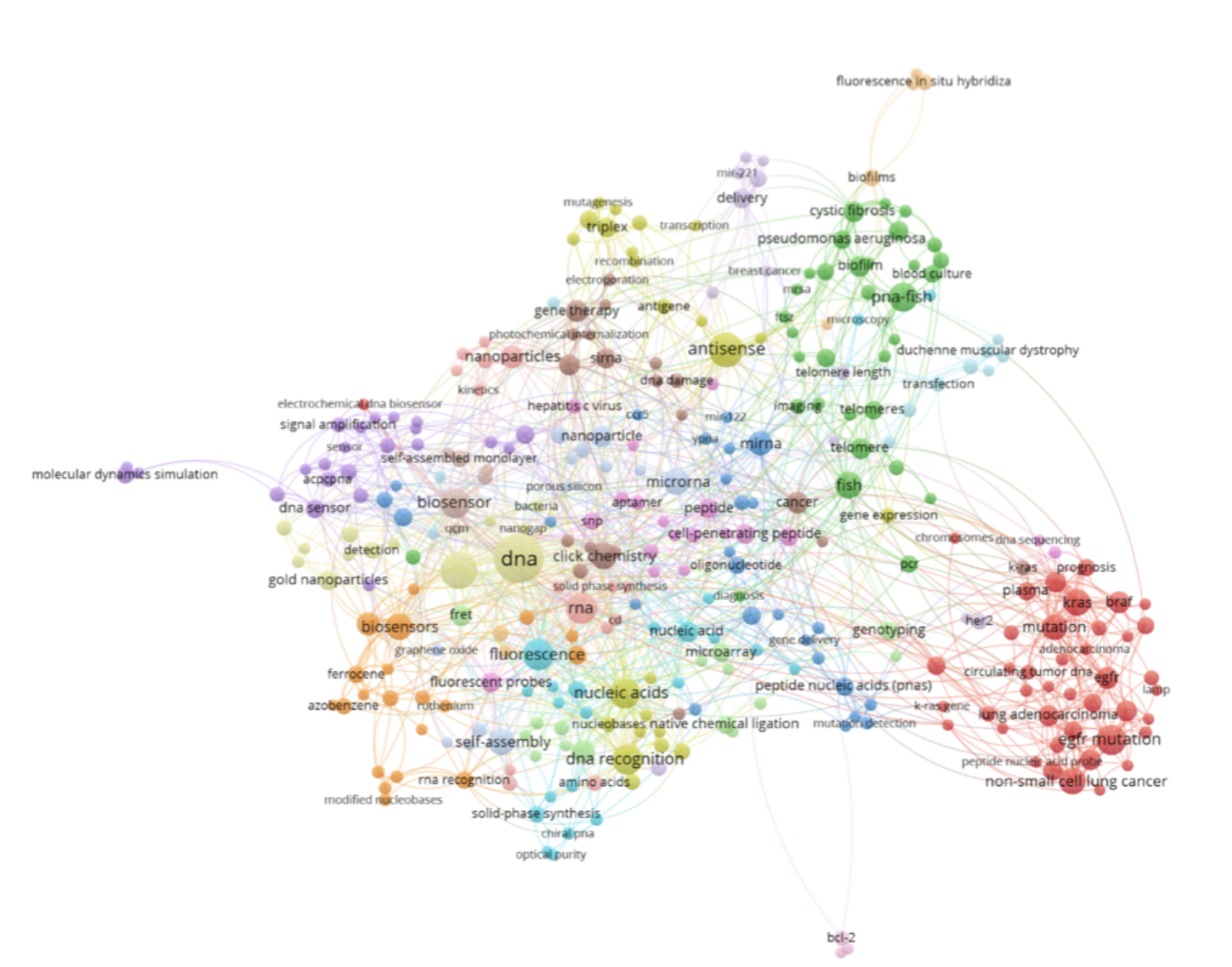
Действительно, программа выделяет больше кластеров, которые мы отобразили, используя для удобства Lin-Log укладку (меню Analysis).
Однако, проблема в том, что авторы используют похожие ключевые слова с вариациями в написании, поэтому на графе можно заметить группы терминов, которые обозначают одно и то же, но попали в разные кластеры (например, biosensorи bisensors, miRNAи microRNA и т.п.). Если мы объединим такие пары, то и разбиение на группы будет выглядеть иначе.
Прежде чем приступить к работе с тезаурусом, познакомимся с данными, которые VOSviewer позволяет выгружать. Кроме само собой разумеющихся иллюстраций в разнообразных форматах (включая векторные EPS, SVG), VOSviewer позволяет сохранять файлы данных, которые можно затем использовать для обработки в других программах (меню Save). Это форматы GML(может быть прочитан в Gephi), Pajek (одноименный софт) и текстовые файлы VOSviewer map и VOSviewer network file.
Рассмотрим подробнее содержимое двух последних файлов:
VOSviewer map file
Файл содержит данные об узлах сети (в нашем случае это термины):
-
Id – идентификатор
-
Label – метка
-
Cluster– номер кластера
-
weight<Links> – количество терминов, вместе с которыми упоминался данный термин
-
weight<Occurrences> – количество статей, в которых упоминался данный термин
-
score<Avg. pub. year> – среднее значение года публикаций
-
score<Avg. citations> – среднее количество цитирований
-
score<Avg. norm. citations> – среднее количество цитирований, нормализованное с учетом возраста статей.
VOSviewer network file
Файл содержит данные о связях между узлами (терминами): первые два столбца относятся к идентификаторам (Id) узлов, в третьем столбце приводится сила связей (которая пропорциональна их количеству).
Таким образом, для тех, кто не владеет навыками программирования, VOSviewer представляет удобнейший сервис для быстрой обработки данных из индексов научного цитирования и реферативных баз, и отражения существующих связей в формате, используемом для анализа сетевых структур.
Работа с тезаурусом
В роли тезауруса VOSviewer использует обычный текстовый файл (.txt) с двумя столбцами – label(термин, который следует заменить) и replace by(термин для замены), разделенных табуляцией.
Для создания его удобно воспользоваться файлом VOSviewer map file, который уже содержит столбец label. Таким образом, нам остается только скопировать этот столбец, выбрать термины, которые следует заменить, добавить рядом второй столбец с названием replace by и заполнить последний вариантами замены.
Это ручной труд, но ориентируясь на столбец Weight<Occurences>, исследователь может ограничиться модификацией лишь наиболее крупных узлов (часто встречающихся терминов), которые оказывают более сильное влияние на разделение кластеров.
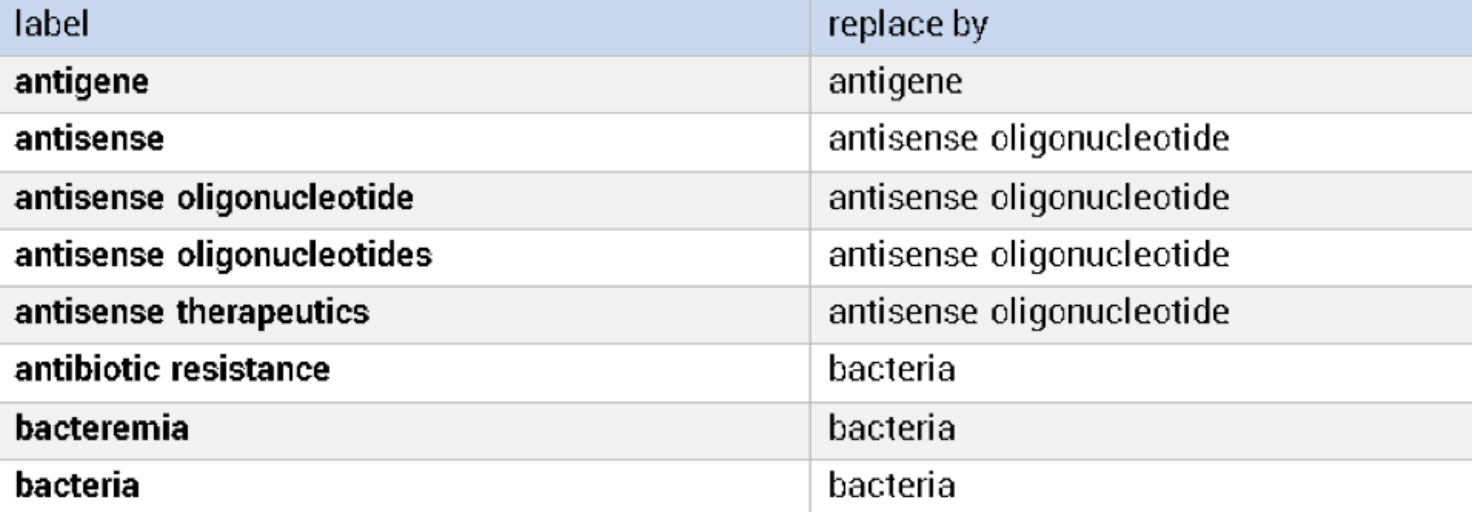
Редакцию тезауруса удобно проводить в программе Excel, поскольку можно будет с помощью фильтра находить варианты однокоренных терминов. В нашем случае, составление тезауруса с вариантами замены для приблизительно 150 терминов заняло 22 минуты.
Загружаем заново файл с данными, указываем VOSviewer на файл тезауруса, сохраняем минимальный порог в 3 упоминания, и видим, что программа обнаруживает уже не 328, а 232 термина – благодаря работе с тезаурусом мы не только унифицировали термины (что окажет положительный эффект на кластеризацию), но и на четверть уменьшили их количество терминов, сделав граф более удобным для визуального прочтения.
Взглянем на результат.

Направления использования пептид-нуклеиновых кислот видны ещё более отчетливо, хотя говорить о качественном выделении отдельных направлений, безусловно, не приходится.
Упорные исследователи могут продолжить работать с тезаурусом и попробовать изменять параметры (можно увеличить Resolutionс 1 до 1.5 - 3), что должно привести к лучшему разделению тем. Любители чтения, вероятно, уже пришли к выводу, что потраченное время лучше было бы пустить на чтение обзоров, а результаты всегда можно представить в виде текста или таблицы.
Часть 4.
В предыдущей части мы использовали VOSviewerдля того, чтобы через анализ ключевых слов выделить отдельные исследовательские направления, связанные с использованием пептид-нуклеиновых кислот. В этот раз мы попытаемся решить эту задачу с помощью другой доступной опции VOSviewer, а именно с помощью анализа названий и рефератов статей.
VOSviewer использует свой собственный алгоритм для выделения существительных (и словосочетаний на их основе), что позволяет увидеть и анализировать более разнообразный набор терминов по сравнению с ключевыми словами.
Шаг 1. запускаем файл VOSviewer.exe
Шаг 2. нажимаем Create
Шаг 3. выбираем Create a map based on text data

Шаг 4. выбираем файл CSV, выгруженный из Scopus
Шаг 5. оставляем выделенными пункты
v Ignore structured abstract labels
v Ignore copyright statements
Шаг 6. Выбираем анализ только названий статей
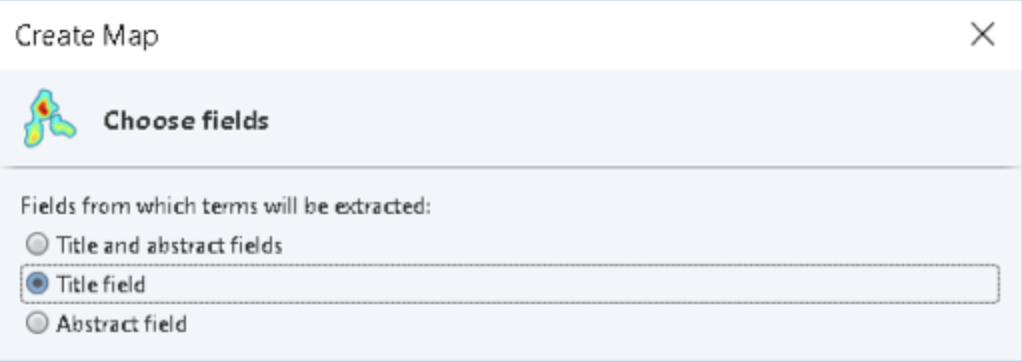
Анализ по названиям удобен для получения быстрой картины, но также предпочтителен, поскольку в названиях, как правило, содержится больше специфических терминов, чем в рефератах.
Шаг 7. выбираем binary counting – тип подсчета, при котором учитывается не количество упоминаний термина внутри названия (или реферата), а только присутствие (1 – если термин встречается, 0 – если термин отсутствует).
На этом же этапе можно указать программе на файл с тезаурусом (см. предыдущий пост), что мы и сделаем.
Шаг 8. Устанавливаем минимальный порог упоминаний на 4, что позволит нам увидеть 260 терминов.
Шаг 9. Не соглашаемся с предложением программы показывать только часть терминов – указываем 260.
Шаг 10. Программа предлагает нам вновь редактировать список терминов, который отфильтрован по убыванию релевантности. Это удобно, поскольку в верхней части таблицы собраны более значимые термины, а в нижней – более общие. В этом списке удаляем всё, что слабо характеризует направление исследований (case report, study, reaction, use, sequence, human cell и т.п.).
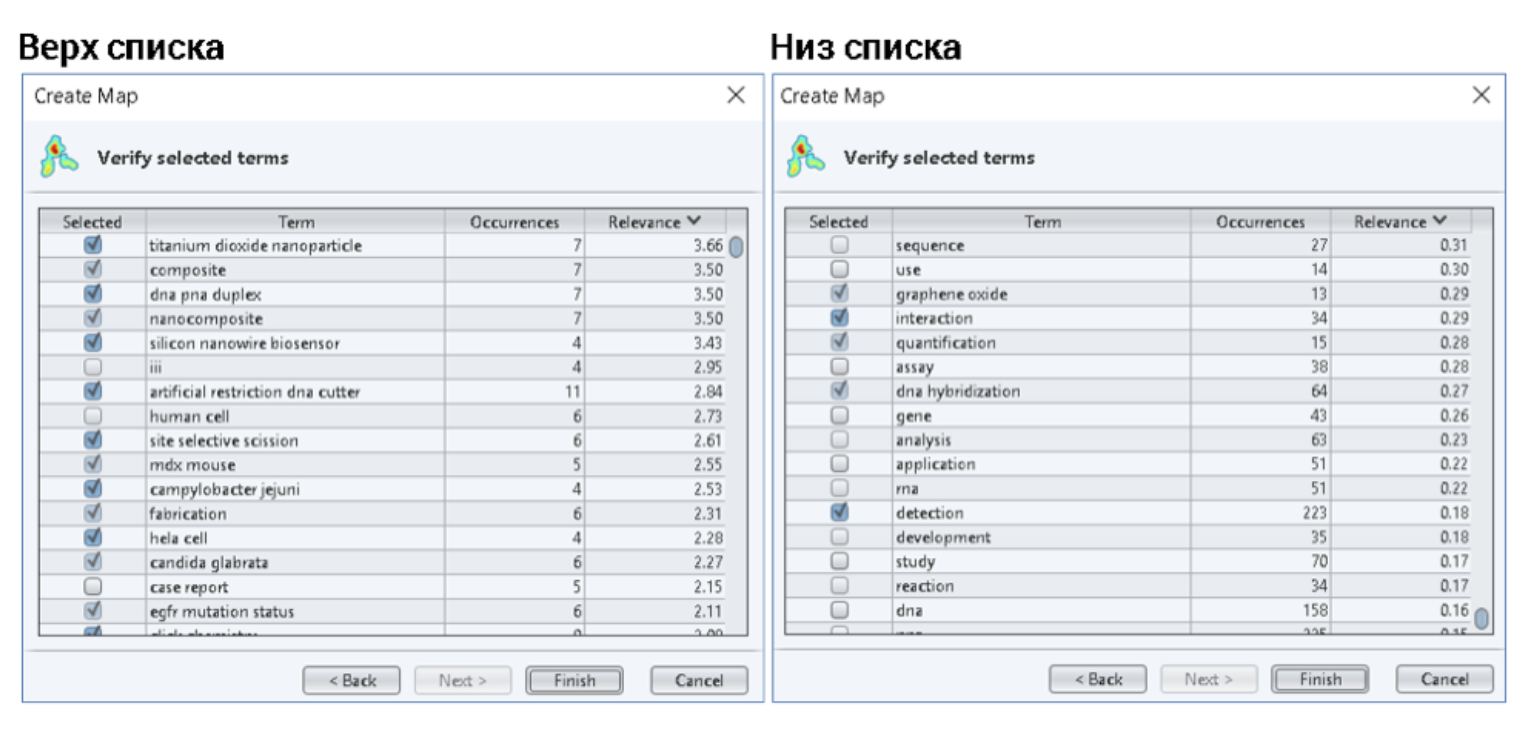
Любуемся результатом.
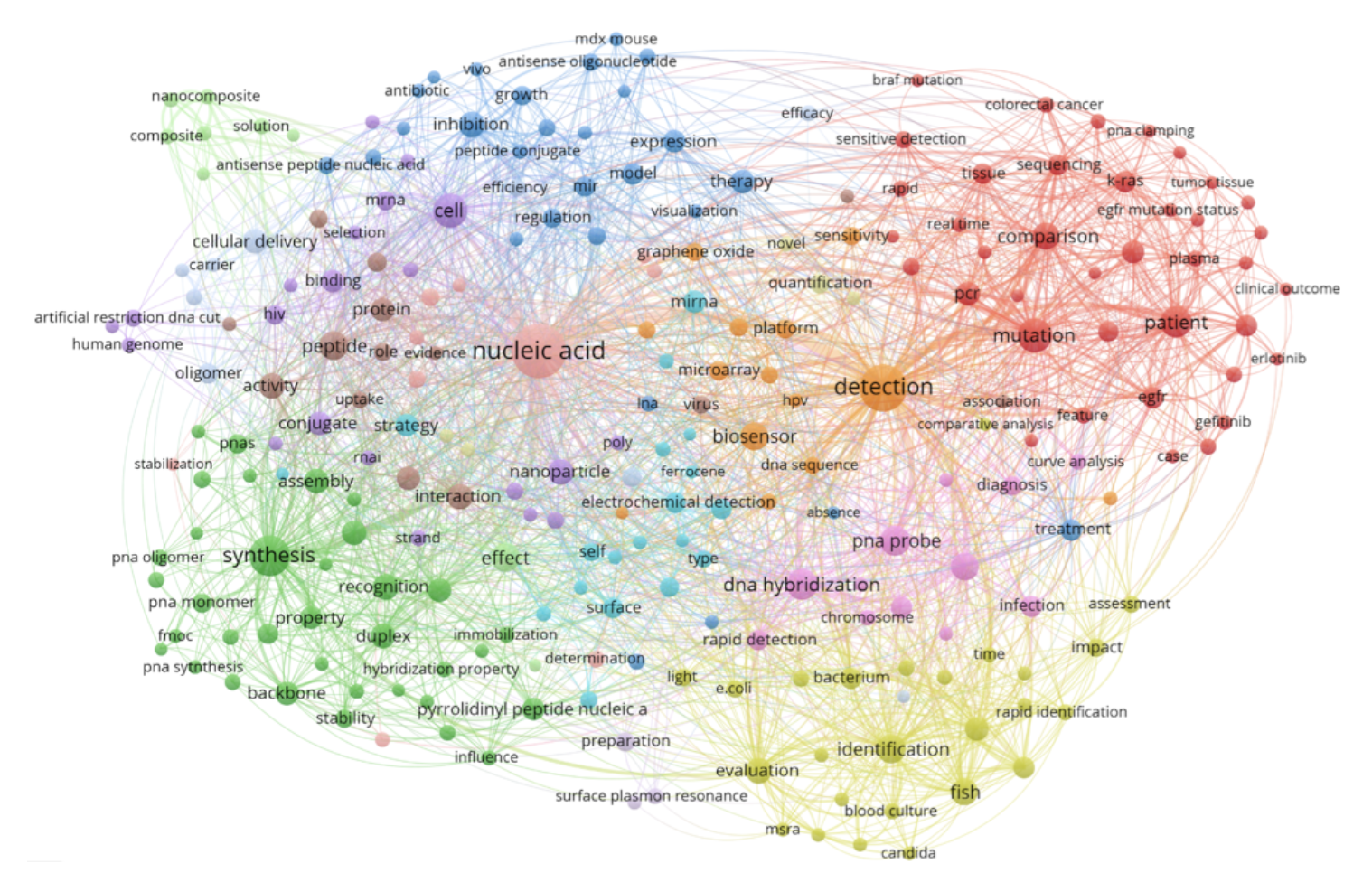
Заключение
Очевидно, что использование VOSviewer для терминологического анализа темы имеет ограничения:
-
во-первых, из-за присутствия синонимов, словоформ и аббревиатур работа с тезаурусом неизбежна
-
во-вторых, программа не позволяет манипулировать отдельными узлами, объединять их в получившемся графе или переносить на отдельный граф, что лишает исследователя возможности сразу увидеть результат задуманных действий – для этого требуется повторно проходить этапы отбора и фильтрации.
В дополнение к этому, программа предлагает ограниченные возможности влиять на укладку графа - выбор пользователя сводится к перебору нескольких алгоритмов. В следующих постах мы рассмотрим программу Gephi, которая имеет несравнимо более богатый функционал для работы с графами.
Однако перечисленные недостатки нивелируются несомненными достоинствами
VOSviewer, к которым следует отнести простоту использования, скорость работы, гибкость в плане загрузки и выгрузки данных, возможность работы с материалами из разных баз данных.
Рассмотренная нами задача и примеры решения являются, безусловно, умозрительными – большинство исследователей вместо составления тезауруса предпочтут найти и прочесть хороший обзор о peptide nucleic acids. Специалисты с навыками программирования назовут готовые «пакетные решения» для популярных языков программирования (R, Python) – более того, будут настаивать на том, что лучше потратить время на изучение этих языков, чем на ручную фильтрацию терминов. И первые, и вторые будут правы – чтение хороших обзоров и освоение навыков программирования не повредят ни одному исследователю.
Однако, автор убежден, что и знакомство с VOSviewer (ну не называть же это «освоением») также имеет смысл – по сочетанию простоты и функциональности эта программа не имеет себе равных в отображении связей внутри публикационных массивов.
Текст сохранен в учебных целях с сайта Эко-вектор.






